コロナ感染者数データに指数曲線あてはめ
タイトルの通り。元データは以下から取得、これは東京都の場合。
* 東京都オープンデータカタログサイト >> 東京都 新型コロナウイルス陽性患者発表詳細
>> https://catalog.data.metro.tokyo.lg.jp/dataset/t000010d0000000068
あてはめた結果が、これ。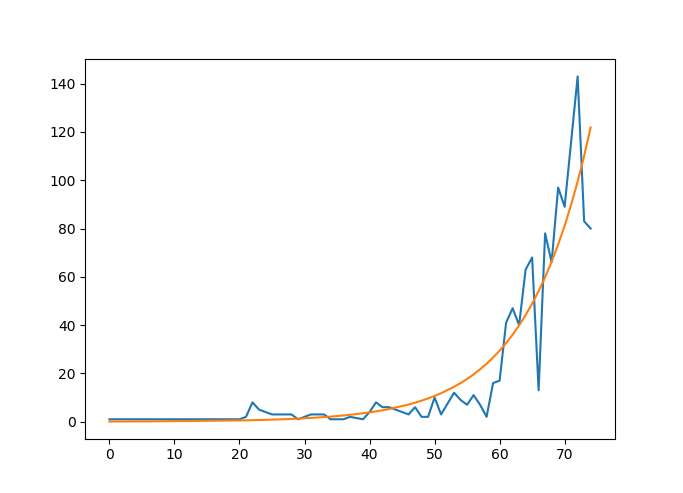
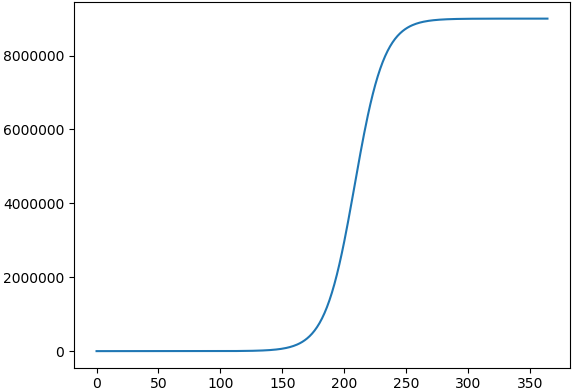
そのまま180日、約半年に期間延長してみる。
★もちろんこれは簡易的なあてはめなので、もっと信頼のおける予測は「SIRモデル」などのキーワードでググってください。
* COVID-19情報共有 >> https://www.fttsus.jp/covinfo/
import numpy as np import pandas as pd import matplotlib.pyplot as plt from scipy.optimize import curve_fit DATA_FILE = "130001_tokyo_covid19_patients.csv" # 読み込むCSVファイル df = pd.read_csv( DATA_FILE ) df = df.rename( columns={"公表_年月日" : "day"} ) # 日本語名をリネームしておこう df['day'] = pd.to_datetime(df.day) # 日付型に変換 df['count'] = 1 # 1行1件とカウントする df = df[ ['day', 'count'] ] # カウント以外は削除 grouped = df.groupby("day").sum() # 日付で集計 x = np.array( grouped.index.map(pd.Timestamp.timestamp) ) # 日付->秒数に変換 base = x[0] # スタートを0日とする day_unit = (24*60*60) # 1日単位に変換する x -= base x /= day_unit y = np.array( grouped['count'] ) # フィットしたい関数 def func( x, a, b ): return np.exp(a * x + b) # 指数関数をあてはめてみよう params, _ = curve_fit( func, x, y ) plt.plot(x, y) plt.plot(x, func(x, *params)) plt.show()
■ 2020/04/15 追記、指数ではなく、ロジスティック曲線をあてはめるには、以下のように差し替える。
# フィットしたい関数 def func( x, a, b ): max_pop = 9000000 # 東京都の最大人口を9百万人とする return ( max_pop / (1 + np.exp( -(a * x + b) ) ) )